|
|
|
| Niveau | Intermédiaire |
| Logiciels utilisés |
QGIS R |
| Plateforme | Windows | Mac | Linux | FreeBSD |
Nous avons vu dans QGIS et géologie structurale: création automatique de rosaces directionnelles et de canevas stéréographiques avec PyQGIS comment créer des scripts Python avec interface dans la Boîte à outils de Traitements (Processing). Il est aussi possible de créer des scripts R de manière comparable. Ce sont des fichiers avec une extension .rsx et ils sont situés dans le dossier /.../.qgis2/processing/rscripts.
Si l’on connait R, ils sont même plus faciles à implémenter.
Principes
Il faut bien entendu que R soit installé et accessible en ligne de commande. Les principes du traitement par le module processing ont été exposés dans QGIS, le cas de l’extension Sextante sur Mac OS X ou comment la connaissance de Python permet de résoudre les problèmes, sans uniquement se lamenter... . Rappelons-les:
- Processing utilise le module Python subprocess pour exécuter directement les commandes R (et pas le module rpy2 qui permet de traiter les commandes R en Python);
- Cela ce fait par un fichier intermédiaire /.../.qgis2/processing/processing_script.r qui est exécuté directement par R, d’où la nécessité que R soit disponible en ligne de commande pour exécuter R processing_script.r
- Si l’on veut utiliser des packages R, il faut donc les installer dans R (ils n’ont rien à voir avec QGIS).
En résumé, le module processing va utiliser directement ce qui est présent dans votre installation de R.
Exemple concret avant les explications: du krigeage « à la ArcGIS »
L’interpolation par krigeage géostatistique est très à la mode chez les utilisateurs d’ArcGIS du fait d'une l’interface conviviale (ou non, voir Du krigeage et du libre et/ou gratuit: inventaire des solutions dans le domaine spatial (logiciels SIGs, applications, librairies, mais sans doute non exhaustif...), même si, pour la plupart, ils ne connaissent que peu de choses sur la géostatistique. Il y a moyen de faire la même chose avec le package automap, vu dans la référence précédente. En pratique, vous lui fournissez des données et il calcule automatiquement le « meilleur » variogramme et sur cette base, il fournit les valeurs de krigeage, de variance et d’erreurs standards en utilisant toutes les arcanes de gstat (rappelons qu'il est toujours possible de décomposer la procédure suivie et de l'adapter si nécessaire).
J’ai été amené à corriger un des scripts qui sont fournis par défaut, Kriging.rsx (voir Bug report #14608: Processing: Kriging rscripts/Kriging.rsx Automap problem and correction )
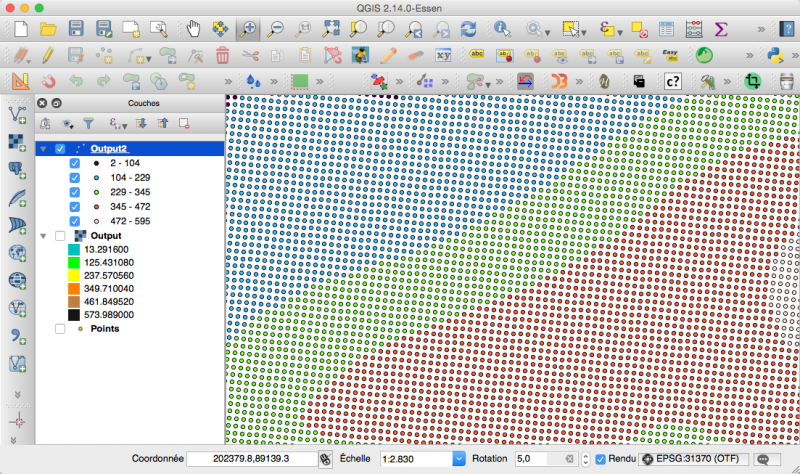
Au départ, je dispose d’une couche de points avec une valeur z dans la table d’attributs:
Je lance mon script R
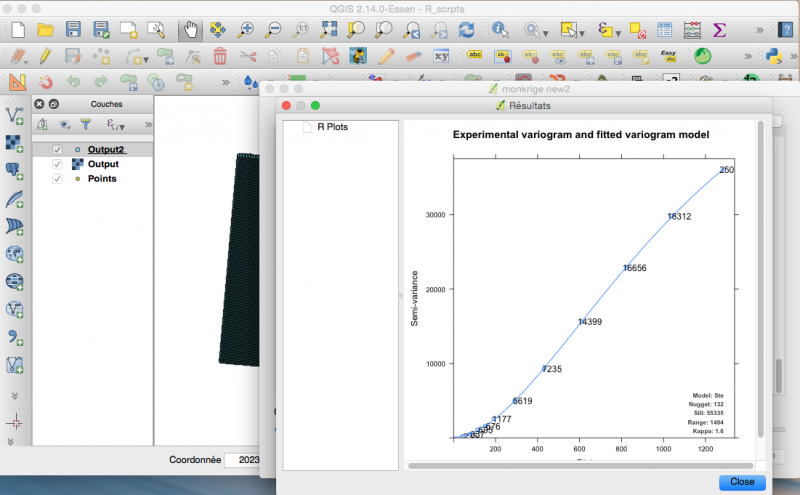
et j’obtiens automatiquement le « meilleur » variogramme
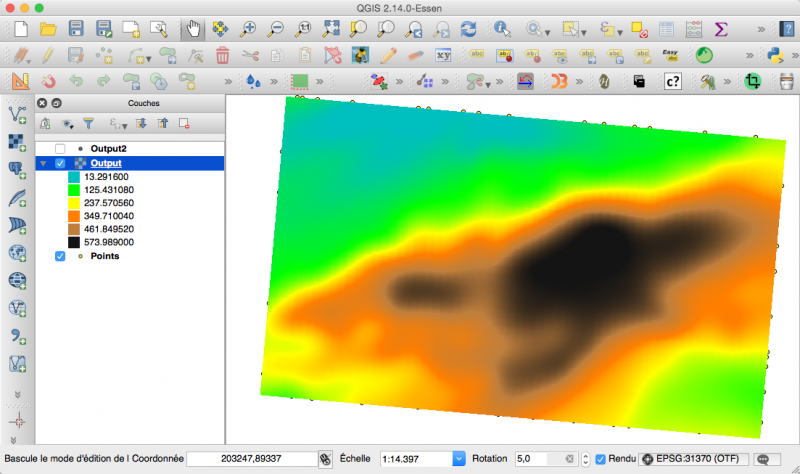
Ainsi que le résultat de krigeage (rasters et points sur une grille)
Explications
Le script n’est pas complexe si l’on connait R
1) création de l’interface
##Basic statistics=group ##showplots ##Layer=vector ##Field=Field Layer ##by=number 0.1 ##Outputr=output raster ##Outputv = output vector
showplot indique que je veux afficher les figures (variogramme), Layer et Field correspondent à la couche et au champ traité, by est la pas de la grille utlisée et Outputr et Outputv les couches raster et vecteur en sortie
2) traitement R
library("automap")
library(raster)
Y<-as.factor(Layer[[Field]])
attribut<-as.data.frame(Y)
A<-as.numeric(Y)
for(j in (1:length(levels(Y))))
for(i in 1:dim(attribut)[1]){
if (attribut[i,1]==levels(Y)[j]){
A[i]=j
}
}
J’utilise les packages automap et raster et je crée la grille d’interpolation
coords<-coordinates(Layer)
MinX<-min(coords[,1])
MinY<-min(coords[,2])
MaxX<-max(coords[,1])
MaxY<-max(coords[,2])
Seqx<-seq(MinX, MaxX, by=by)
Seqy<-seq(MinY, MaxY, by=by)
MSeqx<-rep(Seqx, length(Seqy))
MSeqy<-rep(Seqy, length(Seqx))
MSeqy <- sort(MSeqy, decreasing=F)
Grille <- data.frame(X=MSeqx, Y=MSeqy)
coordinates(Grille)=c("X","Y")
gridded(Grille)<-TRUE
Mesure<- data.frame(LON=coords[,1], LAT=coords[,2],A)
coordinates(Mesure)<-c("LON","LAT")
variogram = autofitVariogram(A~1, Mesure)
plot(variogram)
kriging_result = autoKrige(A~1, Mesure, Grille,model=c("Cir","Lin","Bes","Wav","Hol","Leg","Per","Pen","Mat","Exc","Spl","Ste"))
prediction = raster(kriging_result$krige_output)
3) affichage des couches dans QGIS
L’affichage est automatique (prediction est de la classe RasterLayer de raster) et je transforme les points de la grille avec les valeurs en SpatialPointsDataFrame)
Outputr = prediction Outputv = SpatialPointsDataFrame(kriging_result$krige_output, as.data.frame(kriging_result$krige_output))
Pour aller plus loin
Je vais vous présenter ici quelques traitements supplémentaires sur le raster obtenu par krigeage avec quelques lignes de script pour vous donner l’envie de continuer et d'aller plus loin ...
Je peux représenter mon raster en 3D (pour le plaisir)
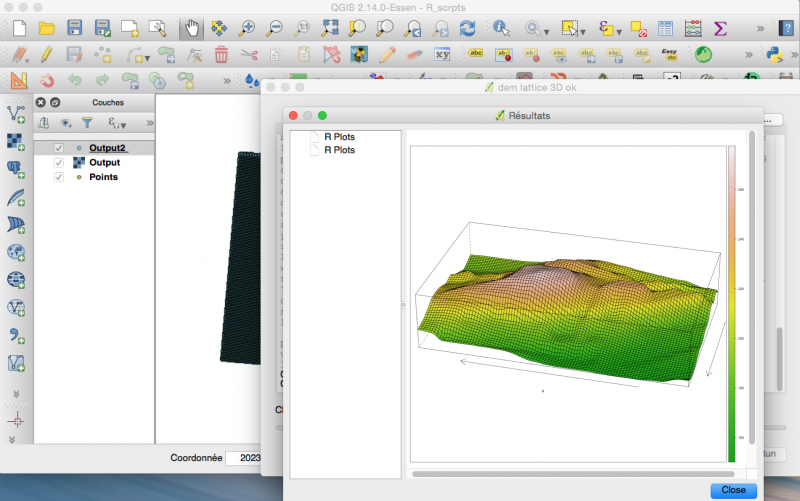
Le script
##Basic statistics=group ##showplots ##Layer=raster dem = Layer dem_df = as.data.frame(dem, xy = TRUE) dem_df = dem_df[complete.cases(dem_df), ] colnames(dem_df)[3] = "z" x_range = diff(range(dem_df$x, na.rm = TRUE)) y_range = diff(range(dem_df$y, na.rm = TRUE)) z_range = diff(range(dem_df$z, na.rm = TRUE)) library(lattice) wireframe(z ~ x * y, data = dem_df,drape = TRUE, colorkey = TRUE,col.regions = terrain.colors(100),screen = list(z = 165, x = -60, y = 0),aspect = c(y_range/x_range, 7*(z_range/x_range)),zoom = 1.1)
Extraire les courbes de niveau
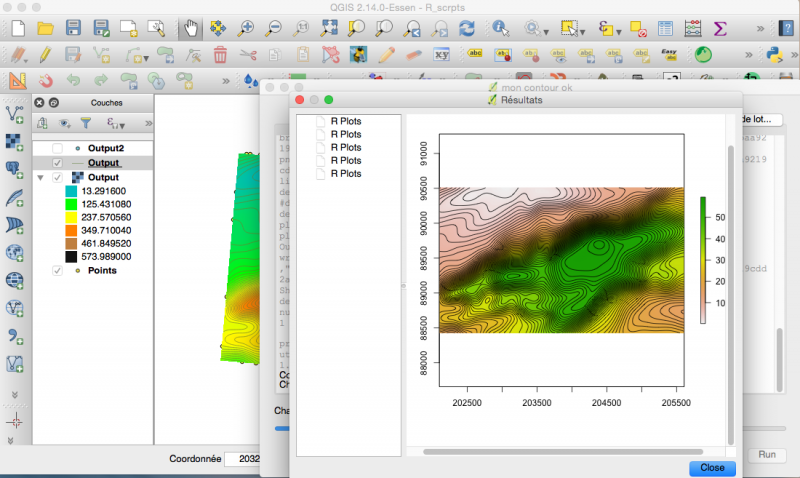

Le script:
##Basic statistics=group ##showplots ##layer=raster ##Output = output vector library(raster) dem <- layer[[1]] dem_contour = rasterToContour(dem, levels = seq(10, 600, 10)) plot(dem) plot(dem_contour, add=TRUE) Output=dem_contour
ou créer un relief ombragé (hillshade)
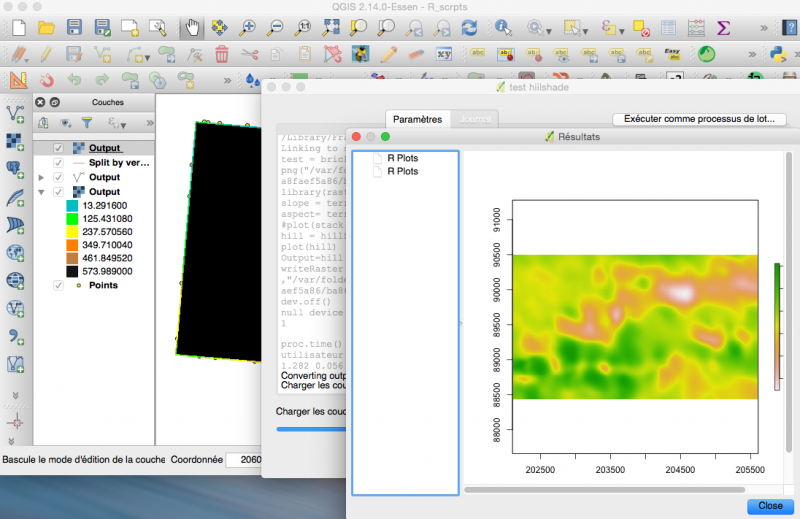
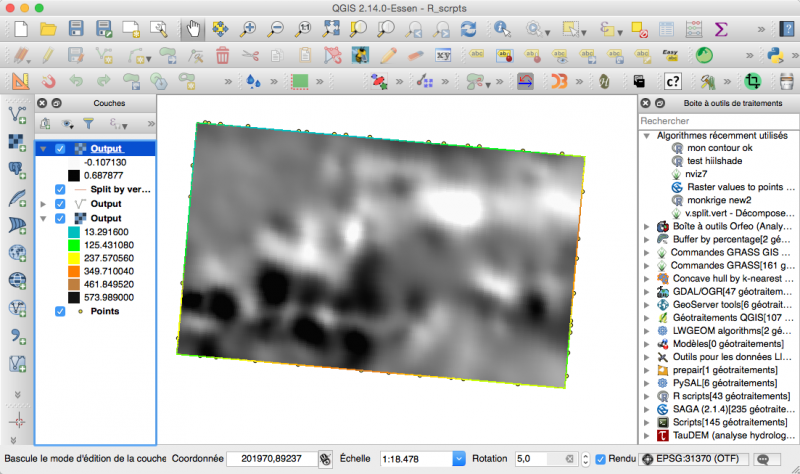
Le script
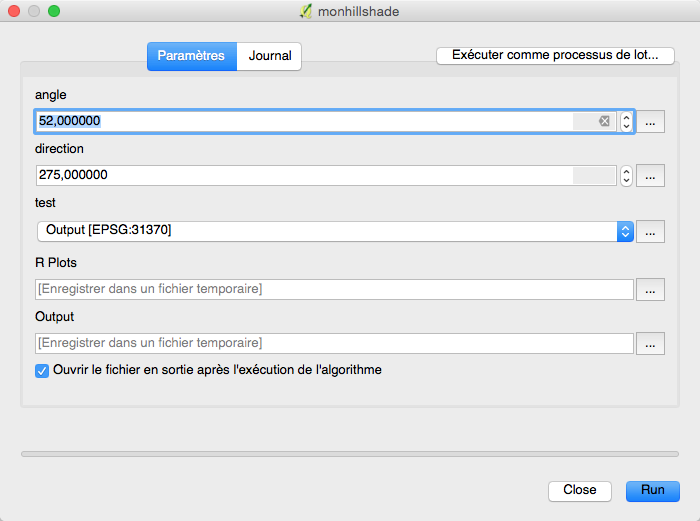
##Basic statistics=group ##showplots ##test=raster ##Output = output raster library(raster) slope = terrain(test,"slope") aspect= terrain(test,"aspect") #plot(stack(slope,aspect)) hill = hillShade(slope,aspect,20,235) plot(hill) Output=hill
Conclusions préliminaires
Il y aurait bien sur moyen d’améliorer tous les scripts comme dans le calcul des courbes de niveau où je fixe les niveaux ou dans le relief ombragé où je fixe l’angle et la direction à l’aide de l’interface (choix des valeurs dans l'interface).
Où trouver des scripts ?
La Boîte à outils dispose d’un script pour créer un nouveau script (R scripts/Outils/Créer un nouveau script) et un autre pour installer les scripts disponibles à QGIS-Processing/rscripts (R scripts/Outils/Obtenir des scripts)
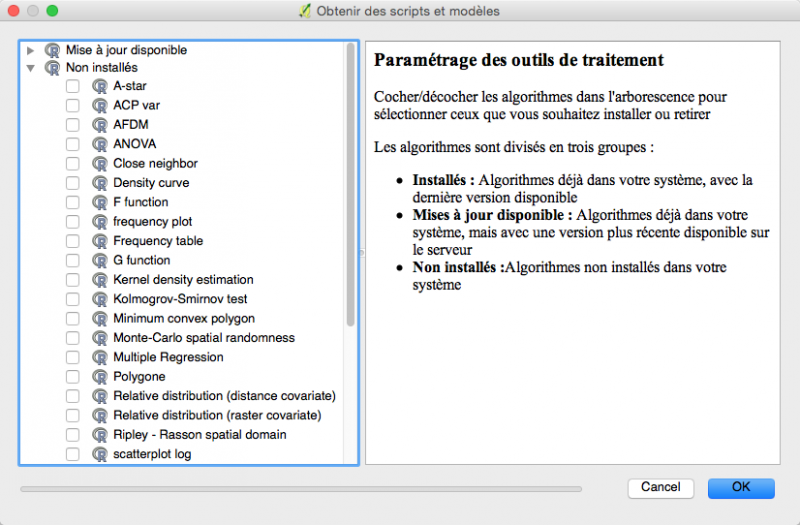
La liste est disponible à list.txt. D’autres scripts spécialisés en géostatistique sont disponibles à INTA SUELOS: QGIS_R_Geostatistics et pour le reste, il faut en trouver ou les créer....
Conclusions
Si vous connaissez R et QGIS, j’espère que ces petits exemples vous donneront des idées pour continuer et aller plus loin. C’est dans l’ensemble beaucoup plus facile qu’avec Python puisque les principaux packages R spatiaux ont déjà tout ce qu’il faut, sans devoir programmer des fonctions équivalentes.
Pour moi, c’est le plus beau traitement du module processing car il ouvre, en particulier, la voie vers des traitements non disponibles dans QGIS ou plus faciles que les traitements des autres Fournisseurs (voir R - spatial : principes généraux et exemples de représentations cartographiques brutes (sans traitements (geo)statistiques) sur le Portail).
Le plus difficile est cependant de convertir les résultats en objets spatiaux R qui sont alors directement affichables dans QGIS. Tous les traitements peuvent être effectués préalablement avec RStudio et si vous observez les scripts disponibles, beaucoup ne font qu'afficher des figures et/ou des résultats comme l'Analyse en Composantes Principales, l'Analyse des Correspondances ou le calcul de diverses valeurs).
Tous les traitements on été faits sur Mac OS X et/ou Linux avec diverses versions de R (3.x.x)
Annexe
Dans le cas du krigeage, le script résultant ( /.../.qgis2/processing/processing_script.r) qui est effectué par R est
> options("repos"="http://cran.at.r-project.org/")
> tryCatch(find.package("automap"), error=function(e) install.packages("automap", dependencies=TRUE))
[1] "/Library/Frameworks/R.framework/Versions/3.2/Resources/library/automap"
> tryCatch(find.package("raster"), error=function(e) install.packages("raster", dependencies=TRUE))
[1] "/Library/Frameworks/R.framework/Versions/3.2/Resources/library/raster"
> tryCatch(find.package("rgdal"), error=function(e) install.packages("rgdal", dependencies=TRUE))
[1] "/Library/Frameworks/R.framework/Versions/3.2/Resources/library/rgdal"
> tryCatch(find.package("raster"), error=function(e) install.packages("raster", dependencies=TRUE))
[1] "/Library/Frameworks/R.framework/Versions/3.2/Resources/library/raster"
> library("raster")
Le chargement a n'ecessit'e le package : sp
> library("rgdal")
rgdal: version: 1.0-4, (SVN revision 548)
Geospatial Data Abstraction Library extensions to R successfully loaded
Loaded GDAL runtime: GDAL 1.11.2, released 2015/02/10
Path to GDAL shared files: /Library/Frameworks/R.framework/Versions/3.2/Resources/library/rgdal/gdal
Loaded PROJ.4 runtime: Rel. 4.9.1, 04 March 2015, [PJ_VERSION: 491]
Path to PROJ.4 shared files: /Library/Frameworks/R.framework/Versions/3.2/Resources/library/rgdal/proj
Linking to sp version: 1.1-1
> Layer = readOGR("/Users/Shared/telechargement/11_04_16/aricle_portail",layer="points_shape")
OGR data source with driver: ESRI Shapefile
Source: "/Users/Shared/telechargement/11_04_16/article_portail", layer: "points_shape"
with 606 features
It has 2 fields
> Field="VALUE"
> by=10
> png("/var/folders/k9/7s3l5fvd5g1dy31b6sms18fr0000gn/T/processing909608e607164409bac78043816115a7/d6d96219f7af4d149e617db7843ff003/RPLOTS.html.png")
> library("automap")
> library(raster)
> Y<-as.factor(Layer[[Field]])
> attribut<-as.data.frame(Y)
> A<-as.numeric(Y)
> for(j in (1:length(levels(Y))))
+ for(i in 1:dim(attribut)[1]){
+ if (attribut[i,1]==levels(Y)[j]){
+ A[i]=j
+ }
+ }
> coords<-coordinates(Layer)
> MinX<-min(coords[,1])
> MinY<-min(coords[,2])
> MaxX<-max(coords[,1])
> MaxY<-max(coords[,2])
> Seqx<-seq(MinX, MaxX, by=by)
> Seqy<-seq(MinY, MaxY, by=by)
> MSeqx<-rep(Seqx, length(Seqy))
> MSeqy<-rep(Seqy, length(Seqx))
> MSeqy <- sort(MSeqy, decreasing=F)
> Grille <- data.frame(X=MSeqx, Y=MSeqy)
> coordinates(Grille)=c("X","Y")
> gridded(Grille)<-TRUE
> Mesure<- data.frame(LON=coords[,1], LAT=coords[,2],A)
> coordinates(Mesure)<-c("LON","LAT")
> variogram = autofitVariogram(A~1, Mesure)
> plot(variogram)
> kriging_result = autoKrige(A~1, Mesure, Grille,model=c("Cir","Lin","Bes","Wav","Hol","Leg","Per","Pen","Mat","Exc","Spl","Ste"))
[using ordinary kriging]
Warning message:
In autofitVariogram(formula, data_variogram, model = model, kappa = kappa, :
Some models where removed for being either NULL or having a negative sill/range/nugget,
set verbose == TRUE for more information
> prediction = raster(kriging_result$krige_output)
> Output<-prediction
> Output2<-SpatialPointsDataFrame(kriging_result$krige_output, as.data.frame(kriging_result$krige_output))
> writeRaster(Output,"/var/folders/k9/7s3l5fvd5g1dy31b6sms18fr0000gn/T/processing909608e607164409bac78043816115a7/2f7b4b596595455db7d3298c6a24ff25/Output.tif", overwrite=TRUE)
> writeOGR(Output2 ,"/var/folders/k9/7s3l5fvd5g1dy31b6sms18fr0000gn/T/processing909608e607164409bac78043816115a7/5bf8086827ee48bda05c5b503554c3a3/Output2.shp","Output2", driver="ESRI Shapefile")
> dev.off()
null device
et dans le fichier qui recense tous les algorithmes effectués (/.../.qgis2/processing/processing.log).
processing.runalg("r:monkrigenew2","/Users/Shared/telechargement/11_04_16/article_portail/points_shape.shp","VALUE",10,None,None,None)
 Site officiel :
Use R scripts in Processing
Site officiel :
Use R scripts in Processing  Site officiel :
CRAN Task View: Mapping tools and services
Site officiel :
CRAN Task View: Mapping tools and services 
licence Creative Commons Paternité-Pas d'Utilisation Commerciale-Pas de Modification 2.0 France

Commentaires
Poster un nouveau commentaire